קובץ:Ensemble quantum 1DOF canonical.png
תוכן הדף אינו נתמך בשפות אחרות.
מראה
מתוך ויקיפדיה, האנציקלופדיה החופשית

גודל התצוגה המקדימה הזאת: 600 × 600 פיקסלים. רזולוציות אחרות: 240 × 240 פיקסלים | 480 × 480 פיקסלים | 900 × 900 פיקסלים.
לקובץ המקורי (900 × 900 פיקסלים, גודל הקובץ: 78 ק"ב, סוג MIME: image/png)
| זהו קובץ שמקורו במיזם ויקישיתוף. תיאורו בדף תיאור הקובץ המקורי (בעברית) מוצג למטה. |
תקציר
| תיאורEnsemble quantum 1DOF canonical.png |
English: Ensemble canonically distributed over energy, for a quantum system consisting of one particle in a potential well. |
| תאריך יצירה | |
| מקור | נוצר על־ידי מעלה היצירה |
| יוצר | Nanite |
Source
. Matplotlib עם נוצרה ה PNG תמונת מפת סיביות
Python source code. Requires matplotlib.
from pylab import *
figformat = '.png'
saveopts = {'dpi':300} #, 'bbox_inches':'tight', 'transparent':True, 'frameon':True}
seterr(divide='ignore')
# Very important number, smaller means more classical (finer-spaced discrete levels, larger means more quantum (fewer discrete levels)
hbar = 0.7/(2*pi)
temp_canonical = 4.1
energy_microcanonical = -2.0
range_microcanonical = 1.0
micro_e0 = energy_microcanonical - 0.5*range_microcanonical
micro_e1 = energy_microcanonical + 0.5*range_microcanonical
def potential(x):
return x**6 + 4*x**3 - 5*x**2 - 4*x
x = linspace(-2.5,2.5,1001)
dx = x[1] - x[0]
U = potential(x)
mass = 1.0
# compute pixel edges, used for pcolormesh.
xcorners = zeros(len(x)+1)
xcorners[:len(x)] = x-0.5*dx
xcorners[-1] = x[-1] + 0.5*dx
# make an energy range, for plots vs energy.
E = linspace(-20,20,10001)
#define color map that is transparent for low values, and dark blue for high values.
# weighted to show low probabilities well
cdic = {'red': [(0,0,0),(1,0,0)],
'green': [(0,0,0),(1,0,0)],
'blue': [(0,0.7,0.7),(1,0.7,0.7)],
'alpha': [(0,0,0),
(0.1,0.4,0.4),
(0.2,0.6,0.6),
(0.4,0.8,0.8),
(0.6,0.9,0.9),
(1,1,1)]}
cm_prob = matplotlib.colors.LinearSegmentedColormap('prob',cdic)
# To get eigenvalues, we need to set up a NxN matrix for the
# Schrodinger equation Hamiltonian. For the momentum operator
# (-hbar^2/(2*m) * d^2/dx^2) the typical central difference
# approximation will be used.
H = zeros((len(x),len(x)))
# set diagonal
H.ravel()[0::len(x)+1] = hbar*hbar/(mass*dx*dx)
H.ravel()[0::len(x)+1] += U
# set above and below diagonal
H.ravel()[1::len(x)+1] = -0.5*hbar*hbar/(mass*dx*dx)
H.ravel()[len(x)::len(x)+1] = -0.5*hbar*hbar/(mass*dx*dx)
# Right, the hamiltonian is set up, so let's just go ahead and
# diagonalize it, poink.
eigval, eigvec = eigh(H)
def doev(H, Emax):
lowE_idx = find(eigval<Emax)
figure()
for i in lowE_idx:
plot(x,eigvec[:,i], label='E = '+str(eigval[i]))
legend(fontsize=8)
micro = ((eigval > micro_e0)*(eigval < micro_e1))*1.0
print "microcanonical (E0 =",energy_microcanonical,", Delta =",0.5*range_microcanonical,") avg energy",
print sum(eigval*micro)/sum(micro)
canonical = exp(-eigval/temp_canonical)
canonical_avgE = sum(eigval*canonical)/sum(canonical)
print "canonical (T =",temp_canonical,") avg energy",
print canonical_avgE
# Boring level plot
fig = figure()
ax = axes()
plot(x,potential(x), linewidth=3)
for i in find(eigval<=13):
axhline(eigval[i], color=(0.5,0.5,0.5),linewidth=0.5,zorder=-1)
ylim(-8,9)
xlim(-2.1,1.7)
fig.get_axes()[0].xaxis.set_ticks([-2,-1,0,1])
xlabel("position $x$")
ylabel("potential $U(x)$")
fig.set_size_inches(3,3)
fig.patch.set_alpha(0)
savefig("quant_potential_eigval_lines"+figformat, **saveopts)
def levelplot(weights):
"""
Plot the potential with eigenstates' wavefunctions superimposed (shown).
weights: list fractions to multiply each eigenstate probability
(e.g., weight 0: do not show. weight 1: fully show)
name: filename to save to
"""
fig = figure()
ax = axes([0.08,0.1,0.73,0.89]) #([0.125,0.1,0.71,0.8])
plot(x,potential(x), linewidth=2, color='r', zorder=-1)
maxp = dx*3.5*amax(weights)
eigwidth = 0.2
for i in find(eigval<=9):
# Here, we plot the eigenfunctions as horizontal bars of varying darkness,
# with height set by the energy eigenvalue.
if weights[i] == 0: continue # don't plot levels with zero weight
pdist = eigvec[:,i]**2 * weights[i]
pdist.shape = (1,len(x))
extent = (amin(x)-0.5*dx, amax(x)+0.5*dx, eigval[i]-0.5*eigwidth, eigval[i]+0.5*eigwidth)
img = imshow(vstack((pdist,pdist)), cmap=cm_prob, extent=extent, interpolation='none', aspect='auto')
# Alternate code using pcolormesh doesn't work because of ugly edges.
# ycorners = vstack([
# [eigval[i]-0.5*eigwidth]*(len(x)+1),
# [eigval[i]+0.5*eigwidth]*(len(x)+1) ])
# pcolormesh(vstack([xcorners,xcorners]), ycorners, pdist, cmap=cm_prob)
clim(0,maxp)
ylim(-9,9)
xlim(-2.1,1.7)
fig.get_axes()[0].xaxis.set_ticks([-2,-1,0,1])
ax.xaxis.set_ticklabels([])
ax.yaxis.set_ticklabels([])
ax.xaxis.labelpad = 2
ax.yaxis.labelpad = -3
xlabel("position $x$")
ylabel("energy")
ax = axes([0.83,0.1,0.14,0.89], axisbg=(0.95,0.95,0.95))
ax.xaxis.set_ticks([])
ax.yaxis.set_ticklabels([])
ax.yaxis.set_ticks_position('right')
ylim(-9,9)
xlabel("states")
dos = E*0.0
for i,Elevel in enumerate(eigval):
# Here we sum up the density of states function
if Elevel > 20: continue # don't waste time with high levels
dos += exp(-4*((E-Elevel)/eigwidth)**2) * weights[i]
fill_betweenx(E, dos, linewidth=0, color=(0.2,0.2,0.76))
xlim(-0.05*max(dos),max(dos)*1.1)
fig.set_size_inches(3,3)
fig.patch.set_alpha(0)
levelplot(ones(len(eigval)))
savefig("quant_potential_eigval_pdists"+figformat, **saveopts)
levelplot(micro)
sca(gcf().axes[0])
axhspan(micro_e0, micro_e1, color=(0.7,1,0.7),zorder=-2)
sca(gcf().axes[1])
axhspan(micro_e0, micro_e1, color=(0.7,1,0.7),zorder=-2)
savefig("quant_potential_eigval_pdists_micro"+figformat, **saveopts)
levelplot(canonical)
sca(gcf().axes[0])
annotate("$\\langle E\\rangle$", (-0.5,canonical_avgE),
textcoords=None,verticalalignment='top',color=(0,0.4,0))
axhline(canonical_avgE, linestyle='dotted', linewidth=1,color=(0,0.4,0))
annotate('',(1.2,7.-temp_canonical),(1.2,7.),
arrowprops = {'arrowstyle':'<->'})
text(1.15,7.-0.5*temp_canonical,'$kT$',
horizontalalignment='right',verticalalignment='center')
sca(gcf().axes[1])
axhline(canonical_avgE, linestyle='dotted', linewidth=1,color=(0,0.4,0))
fill_betweenx(E, exp(-E/temp_canonical), linewidth=0, color=(0.7,1,0.7),zorder=-2) # green exponential
savefig("quant_potential_eigval_pdists_canonical"+figformat, **saveopts)
# Position expectation values
figure()
pdist = zeros(len(x))
for i,p in enumerate(micro): pdist += p*eigvec[:,i]**2
if any(micro):
plot(x, pdist/sum(micro)/dx, label='microcanonical')
pdist = zeros(len(x))
for i,p in enumerate(canonical): pdist += p*eigvec[:,i]**2
plot(x, pdist/sum(canonical)/dx, label='canonical', color='g')
xlim(-2.1,1.7)
fig.get_axes()[0].xaxis.set_ticks([-2,-1,0,1])
xlabel("position $x$")
ylabel("PDF of position $P(x)$")
legend()
savefig("quant_position_pdf"+figformat, **saveopts)
רישיון
אני, בעל זכויות היוצרים על עבודה זו, מפרסם בזאת את העבודה תחת הרישיון הבא:
| קובץ זה זמין לפי תנאי הקדשה עולמית לנחלת הכלל CC0 1.0 של Creative Commons. | |
| האדם ששייך יצירה להיתר הזה הקדיש את היצירה לנחלת הכלל על־ידי ויתור על כל הזכויות שלו או שלה על היצירה בכל העולם לפי חוק זכויות יוצרים, לרבות כל הזכויות הקשורות או הסמוכות כקבוע בחוק. באפשרותך להעתיק, לשנות, להפיץ, או להציג את היצירה, אפילו למטרות מסחריות, וכל זה אפילו מבלי לבקש רשות.
http://creativecommons.org/publicdomain/zero/1.0/deed.enCC0Creative Commons Zero, Public Domain Dedicationfalsefalse |
כיתובים
נא להוסיף משפט שמסביר מה הקובץ מייצג
פריטים שמוצגים בקובץ הזה
מוצג
ערך כלשהו ללא פריט ויקינתונים
30 באוקטובר 2013
היסטוריית הקובץ
ניתן ללחוץ על תאריך/שעה כדי לראות את הקובץ כפי שנראה באותו זמן.
| תאריך/שעה | תמונה ממוזערת | ממדים | משתמש | הערה | |
|---|---|---|---|---|---|
| נוכחית | 00:51, 31 באוקטובר 2013 | 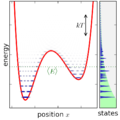 | 900 × 900 (78 ק"ב) | Nanite | User created page with UploadWizard |
שימוש בקובץ
![]() אין בוויקיפדיה דפים המשתמשים בקובץ זה.
אין בוויקיפדיה דפים המשתמשים בקובץ זה.
שימוש גלובלי בקובץ
אתרי הוויקי השונים הבאים משתמשים בקובץ זה:
- שימוש באתר en.wikipedia.org
- שימוש באתר fa.wikipedia.org
- שימוש באתר fr.wikipedia.org
מטא־נתונים
קובץ זה מכיל מידע נוסף, שכנראה הגיע ממצלמה דיגיטלית או מסורק שבהם הקובץ נוצר או עבר דיגיטציה.
אם הקובץ שונה ממצבו הראשוני, כמה מהנתונים להלן עלולים שלא לשקף באופן מלא את הקובץ הנוכחי.
| רזולוציה אופקית | 118.11 dpc |
|---|---|
| רזולוציה אנכית | 118.11 dpc |
